3章 Fitness landscape and Sequence spaces
3.1 sequence space
長さLのsequenceがあったときに、有り得るsequenceは通り。(A,T,C,G)
このL次元空間をsequence spaceとする。
これを2進数(0,1)で考えると、L次元の超立方体の格子点があり得る組み合わせとなる。
各格子点の距離はマンハッタン距離で考えられる。
3.2 Fitness landscape
Fitness landscapeはsequence space状の格子点に対し、数値(reproduction rate)を与えたもの。
各格子点の評価みたいなもの。
3.3 the equasispecies equation
2進数でのsequence spaceを考え、それぞれの格子点を10進数に変換すると0からまで1対1で配列番号として与える。
i番目の配列に対して、をそれぞれiの割合、適応度、配列jからiへの変異行列とすると、
ただし、
をequasispecies equationという。
Qが単位行列、つまり変異が起きないなら最も高い をもつ配列iが生き残る。
Qが単位行列でないなら、単体内のある点 に収束する。このときφは必ずしも最大値とはならない。
しかし、総個体数はφの指数関数で増加する。
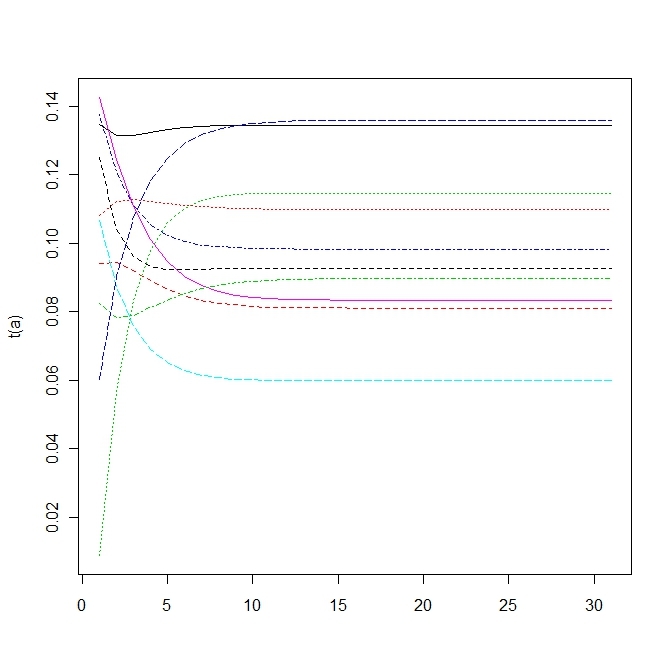
#試行回数
t<-30
#変異しうる配列数
n<-10
#遺伝子iを持つ個体の割合ベクトル
x<-runif(n)
x<-x/sum(x)
#遺伝子iを持つ個体のfittnessベクトル
f<-runif(n)
#mutation matrix
Q<-matrix(runif(n*n),n,n)
q<-apply(Q,1,sum)
Q<-Q/q
#average fittness
phi<-x%*%f
a<-matrix(0,n,t+1)
a[,1]<-x
for(i in 1:t){
dx<-(x*f)%*%Q-phi%*%x
x<-x+dx
a[,i+1]<-x
phi<-x%*%f
}
matplot(t(a),type="l")3.4 a mutation matrix for point mutations
配列i,jのハミング距離をとして、点変異の入る確率をuとすると
変異行列Qの各成分(iからjと変異する確率)は
3.5 a adaptation is localization in sequence space
変異する確率が高すぎると適応していた個体(fittnessの値が高い)の割合がいまいち増えない。
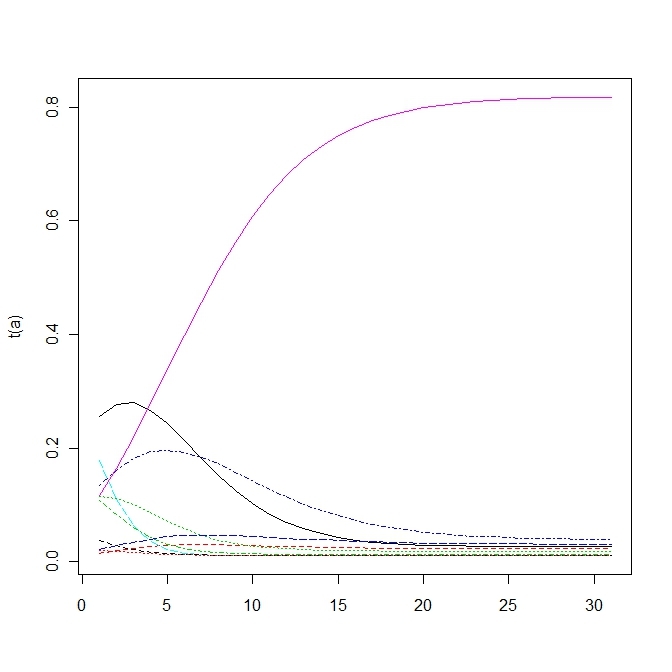
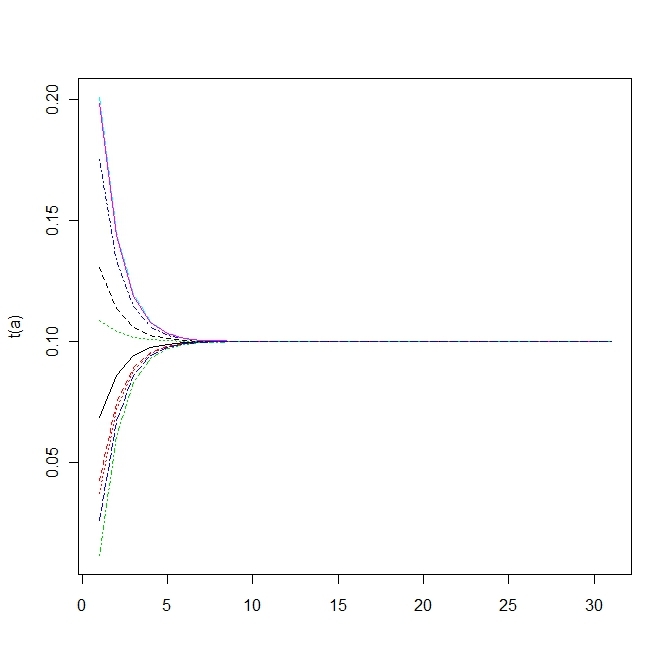
上の方は変異確率を1/100にしたもので、下の方は1/10にしたもので、上だともっとも高いfittnessをもつものに収束する。
単純なモデルでこの変異の閾値を計算すると という条件が求められる。
つまりで、RNAウイルス、大腸菌、酵母菌、マウス、人などでuLの実測値が1より低いという論文が出ているらしい(Drake(1991,1998))。
一方で、QβウイルスとVZVウイルスは1よりはるかに大きく、その理由は不明。
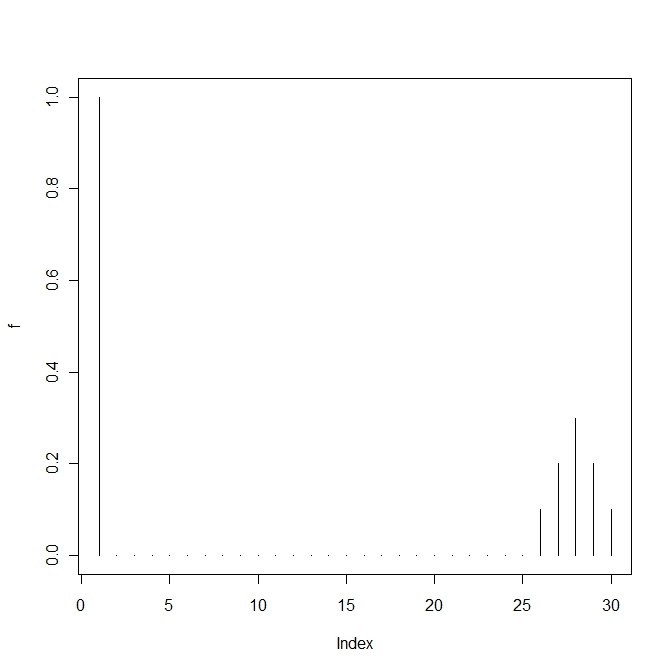
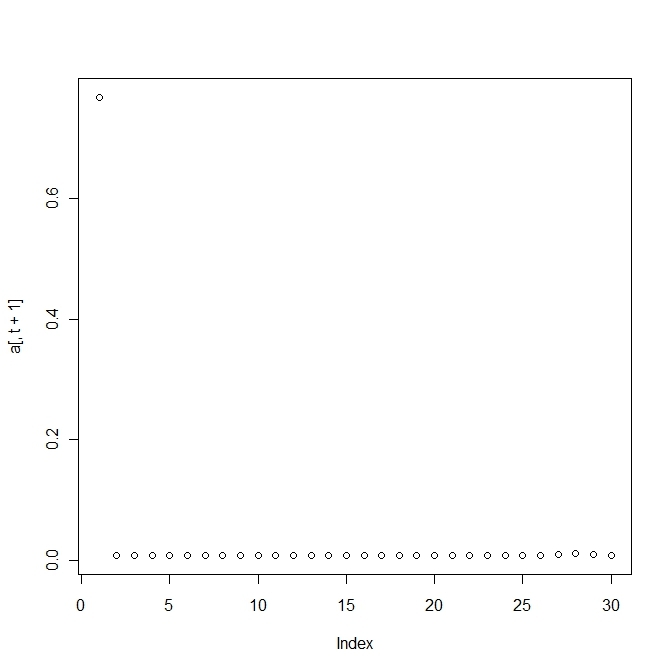
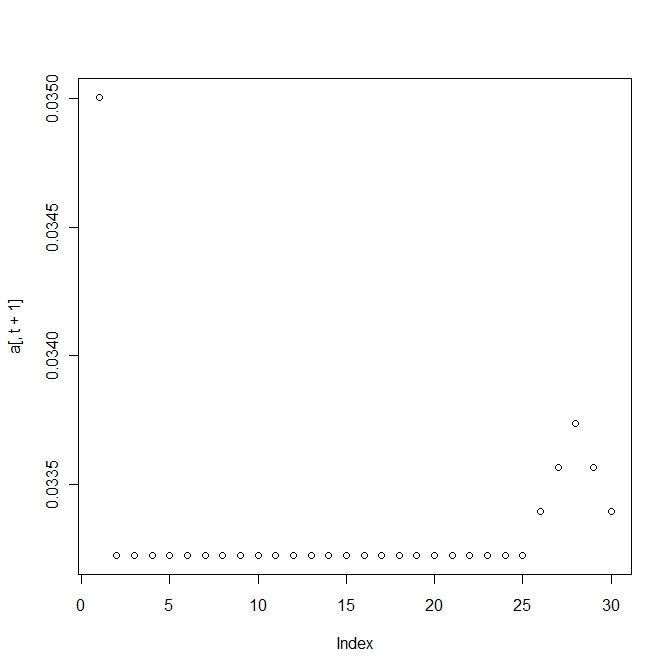
3.6 selection of the quasispecies
2峰性のfittness landscapeをもち、1つは高いが狭く、1つは高くはないが幅広とする。
このとき、変異の割合によって生き残りが変わってくる。
変異の割合が高すぎればすべての種が生き残り、低すぎると最も高いものに収束する。
その中間であれば、2つのピークをもつ種が生き残る。
#試行回数
t<-100
#変異しうる配列数
n<-30
#遺伝子iを持つ個体の割合ベクトル
x<-runif(n)
x<-x/sum(x)
#遺伝子iを持つ個体のfittnessベクトル
f<-c(1,rep(0,n-1))+c(rep(0,n-5),0.1,0.2,0.3,0.2,0.1)
#mutationの割合を調整するパラメーター
h<-100
#mutation matrix
Q<-matrix(1,n,n)+h*diag(n)
q<-apply(Q,1,sum)
Q<-Q/q
#average fittness
phi<-x%*%f
a<-matrix(0,n,t+1)
a[,1]<-x
for(i in 1:t){
dx<-(x*f)%*%Q-phi%*%x
x<-x+dx
a[,i+1]<-x
phi<-x%*%f
}
plot(a[,t+1])